26. August 2020
Neue Erkenntnisse über den Türöffner von SARS-CoV-2
Das Spikeprotein auf der Virusoberfläche ist unerwartet beweglich
Die Suche nach einem geeigneten Impfstoff ist ein zentrales Forschungsgebiet im Kampf gegen das Coronavirus SARS-CoV-2. Ein wichtiger Schritt hierfür ist es zu verstehen, wie das Virus in die Zellen eindringt. Verantwortlich dafür ist ein Protein. Als „Türöffner“ sitzt das SARS-CoV-2 Spike (S)-Protein auf der Virusoberfläche und sorgt dafür, dass das Virus in die Wirtszelle eintreten kann.
Um die Entwicklung eines schützenden Impfstoffs zu erleichtern verwenden die FIAS Wissenschaftler Gerhard Hummer und Roberto Covino fortschrittliche Computersimulationen, um Schwachstellen auf der Oberfläche des S-Proteins zu identifizieren, die von Antikörpern angegriffen werden könnten. Im Verbund mit Wissenschaftlerinnen und Wissenschaftlern des Max-Planck-Instituts für Biophysik, dem EMBL, dem Paul Ehrlich Institut (PEI) und weiteren Forschungseinrichtungen haben sie das Spikeprotein umfassend analysiert und modelliert. In der Onlineausgabe von Science vom 18.08.2020 berichten sie über ihre Forschung und die Erkenntnis, dass das Spikeprotein deutlich beweglicher ist, als zuerst angenommen.
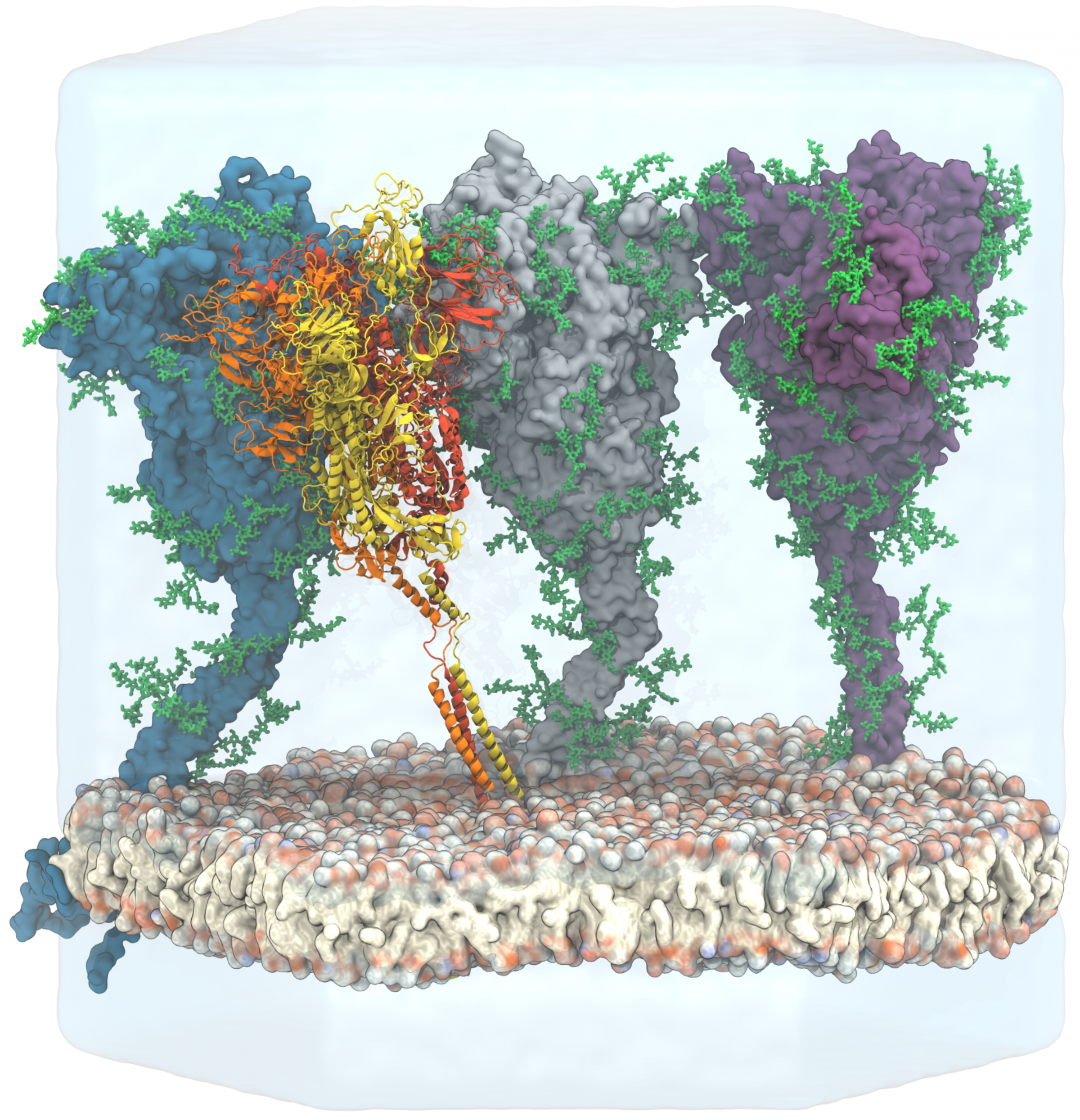
Durch ihre vielseitige Expertise konnte das Protein sowohl mit bildgebenden Verfahren, als auch mit Computersimulationen in seiner natürlichen Umgebung analysiert werden. Um die Dynamik und die Struktur des Spikes zu bestimmen, haben die Biophysiker und Gerhard Hummer und Roberto Covino mit ihren Kollegen vom MPI für Biophysik Modelle aus der Membrandynamik auf das Protein angewandt. Gleichzeitig haben Forschende des PEI SARS-CoV-2-Partikel aus dem Überstand infizierter Zellen gewonnen, welche von den Wissenschaftlerinnen und Wissenschaftlern des EMBL und MPI mithilfe von Kryo-Elektronenmikroskopie in situ (am Virus) genauestens kartiert werden konnten. Diese 3-dimensonalen Bilder des Proteins auf der Oberfläche des Virus wurden daraufhin mit dem theoretischen Modell der Frankfurter Gruppe integriert, um die Struktur und die Dynamik des Proteins im Detail aufzuklären.
Aus den Modellen und Bildern konnten die Forschenden schließen, dass das Protein viel flexibler ist, als vorher angenommen. Diese Eigenschaft ermöglicht es dem Virus, sich wie ein Ankerhaken an menschliche Zellen anzudocken und in sie einzudringen. Zusätzlich hat die interdisziplinäre Kollaboration zuckerähnliche Ablagerungen auf dem Protein entdeckt. Die sogenannten Glykane verstecken die Proteinstruktur vor dem Immunstem und erschweren es den Antikörpern am Protein anzudocken. Diese Informationen sind wichtig, um die Wirksamkeit von Impfstoffen oder Antikörpertherapien einschätzen zu können.
Weitere Informationen:
Publikation bei Science: https://science.sciencemag.org/content/early/2020/08/17/science.abd5223
Pressemitteilung des Paul-Ehrlich Instituts: https://www.pei.de/DE/newsroom/pm/jahr/2020/14-neue-erkenntnisse-spikeprotein-sars-cov-2.html
Bild: Sikora et.al; Map of SARS-CoV-2 spike epitopes not shielded by glycans Lizenz: Creative Commons CC-BY-NC-ND 4.0