4. Juni 2025
Wenn die Bilder laufen lernen: Neue Methode stellt Molekülbewegung dar
Simulationen und Maschinelles Lernen ermöglichen die Auswertung elektronen-mikroskopischer Bilder in Millisekunden
Die für die Funktion von Molekülen wichtige Bewegung besser zu verstehen erlaubt eine neue Methode. Forschende am FIAS entwickelten gemeinsam mit einem Team vom New Yorker Flatiron Institute (USA) eine Technik, um elektronenmikroskopische Bilder mit Hilfe physikbasierter Simulationen und maschinellem Lernen schneller und zuverlässiger auszuwerten. Dies ermöglicht künftig ein besseres Verständnis der Funktionen des Körpers.
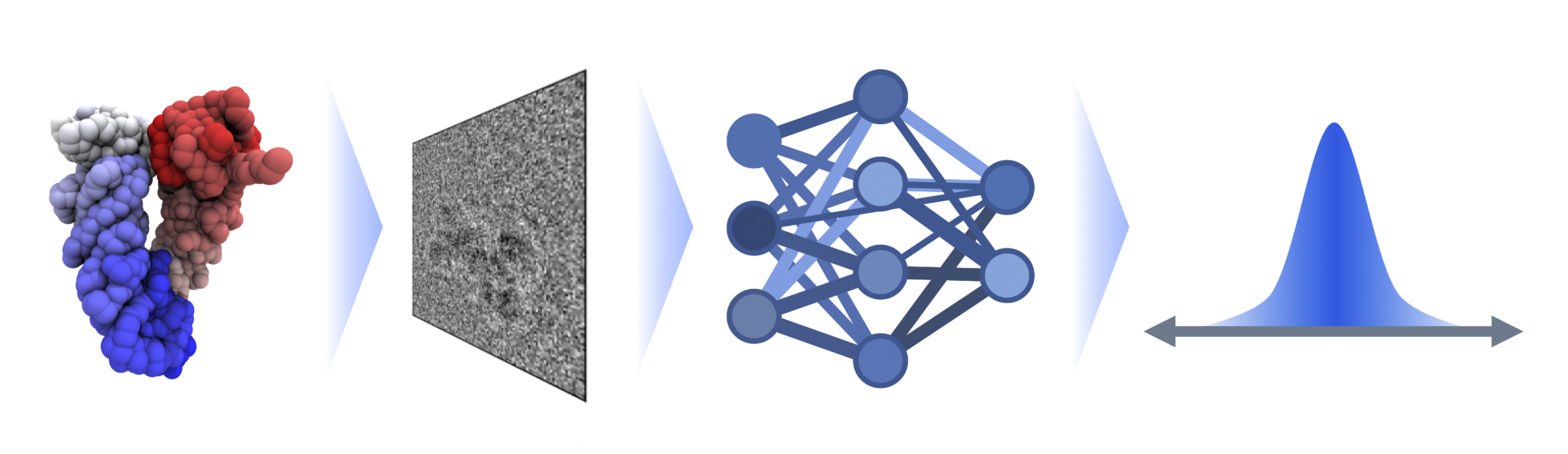
Biologische Moleküle wie Proteine oder DNA sind flexibel und beweglich – dies ist entscheidend für ihre Funktion. Aufnahmen in modernen Elektronenmikroskopen geben jedoch nur statische Bilder wider: In der Kryo-Elektronenmikroskopie werden die Moleküle eingefroren und in dieser starren Struktur fotografiert. „Die einzelnen Aufnahmen dieser winzigen Strukturen sind häufig verrauscht – und wir wissen nicht, von welcher Seite das Molekül aufgenommen ist“, beschreibt FIAS-Doktorand Lars Dingeldein die problematische Auswertung solcher Bilder. Die Gruppe unter Leitung von FIAS-Senior-Fellow Roberto Covino hat daher eine Methode entwickelt, die sowohl den Aufnahmewinkel als auch die Veränderung der Moleküle berechnet.
Dingeldein vergleicht das mit Schnappschüssen von einem Läufer: „Um die Bewegung zu verstehen, benötigt man viele Einzelbilder“. Die Wissenschaftler nutzten daher Biomoleküle mit bekanntem Bewegungsprofil, um ihr System zu trainieren: „Wir simulieren den Mikroskopierprozess und erhalten einen Datensatz von künstlichen Bildern, von denen wir das genaue Aussehen kennen“, so Dingeldein. Damit lernt das System die Struktur eines Biomoleküls anhand der Bilder zu erkennen. Das maschinelle Lernen basiert also auf bekannten Daten – von der Struktur zum Bild – und kann aus diesen Erfahrungswerten später umgekehrt aus Bildern die Struktur errechnen.
Der 27jährige Biophysiker entwickelte mit Hilfe der riesigen Rechenkapazitäten den dafür nötigen Algorithmus. Nachdem das System entsprechend trainiert war, wendeten die Forschenden den Algorithmus auf echte Datensätze an, von denen das Ergebnis bekannt war. Die mit der neuen Methode berechnete Struktur und Bewegung deckte sich mit den bekannten Daten. Die von den FIAS-Forschenden entwickelte Cryo-EM simulationsbasierte Inferenz (cryoSBI) ermöglicht ein deutlich schnelleres Verständnis von Protein- und Genstrukturen: Mit klassischen Methoden dauert es derzeit etwa 15 Sekunden, aus einem einzelnen Bild die Form eines Proteins zu bestimmen. Mit cryoSBI sind es nur noch Millisekunden. „Das Trainieren ist aufwändig, aber hinterher ist die Methode deutlich kostengünstiger“, so Dingeldein. Interessierten Anwenderinnen und Anwendern steht die Technik ab sofort zur Verfügung. „Damit werden wir biologische Systeme schneller und besser verstehen“, ist Gruppenleiter Covino überzeugt. „Etwa für medizinische Anwendungen ist es wichtig, die Bewegung und mögliche krankhafte Störung von Biomolekülen zu verstehen.“ Das Team wird künftig die Methode weiter optimieren und in der Anwendung verbessern.
Publikation: Lars Dingeldein, David Silva-Sánchez, Luke Evans: Amortized template matching of molecular conformations from cryoelectron microscopy images using simulation-based inference, PNAS 122 (23) e2420158122, https://doi.org/10.1073/pnas.2420158122